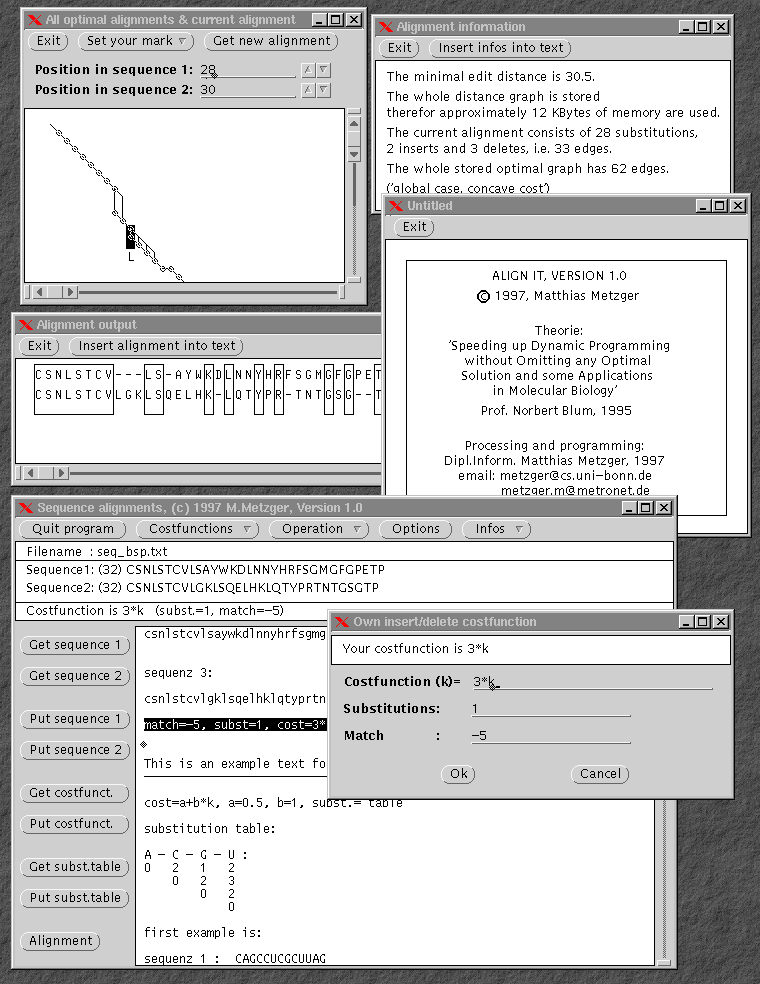
Alignment-Programm:
Aufgabe ist die Berechnung von optimalen Alignments zweier RNA-, DNA- oder Protein- Sequenzen. Der Schwerpunkt besteht darin, alle optimalen Überführungen der beiden eingegebenen Sequenzen zu berechnen und in geeigneter Weise anzuzeigen. Außerdem soll als Zeitschranke für die Berechnungen O(n*m*log(n+m)) (n:=Länge von Sequenz 1, m:=Länge von Sequenz 2) und als Platzschranke O(n*m) nicht überschritten werden.
Folgende Berechnungsmöglichkeiten sind zunächst geplant und auch schon verfügbar:
- Berechnung von globalen Alignments, beliebige Kostenfunktionen für Insert/Delete Operationen
- Berechnung von globalen Alignments, biologisch relevante ('konkave') Kostenfunktionen
- Finde lokale optimale Alignments zwischen einer kurzen und einer langen Sequenz, 'konkave' Kostenfunktionen
- Finde optimale Subalignments zweier Sequenzen, 'konkave' Kostenfunktionen
Um eine einfache Bedienung zu gewährleisten, ist das Programm in eine ansprechende Oberfläche integriert. Es steht für Sun- Station und PC zur Verfügung, die unter UNIX/LINUX mit X Window betrieben werden.
Download:
(Nach dem "download" ggf. 'chmod u=rwx align_it.exe' im entsprechenden Verzeichnis eintippen, um das Programm ausführbar zu machen!)
Bildschirmausschnitt des Programms: